Kamimoto Lab at Osaka University
Publications
Highlights
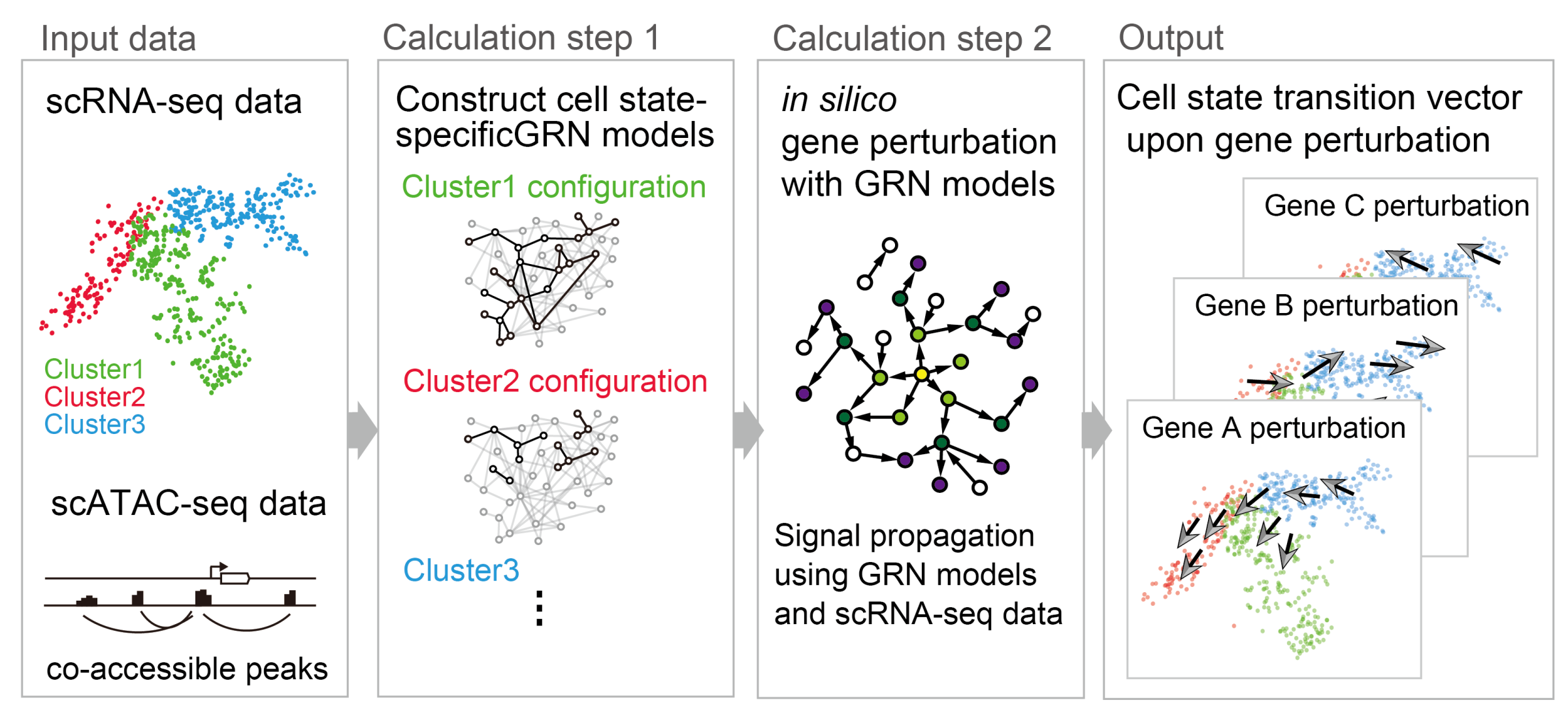
1細胞RNA-seqデータ、1細胞ATAC-seqデータ、転写因子のDNA結合情報などを統合して遺伝子発現制御ネットワークをモデル化する技術CellOracleを開発しました。CellOracleでは作成した遺伝子制御モデルを細胞の予測シミュレーターとして利用するよう設計されており、遺伝子を変動させたときの細胞分化への影響を予測する、in silico gene perturbationを行います。
図は血球分化の過程で重要な転写因子を阻害したときのシミュレーション結果を示します。
複雑な遺伝子ネットワークモデルを予測シミュレーターとして活用することで細胞運命制御における遺伝子機能の直観的な解釈を可能にします。
Kamimoto, K., et al.,
Dissecting cell identity via network inference and in silico gene perturbation.
Nature 614, 742–751 (2023).
本論文はNature のNews & Views で特集されました。Farrel, JA., An oracle predicts regulators of cell identity. Nature 614, 630-632 (2023)
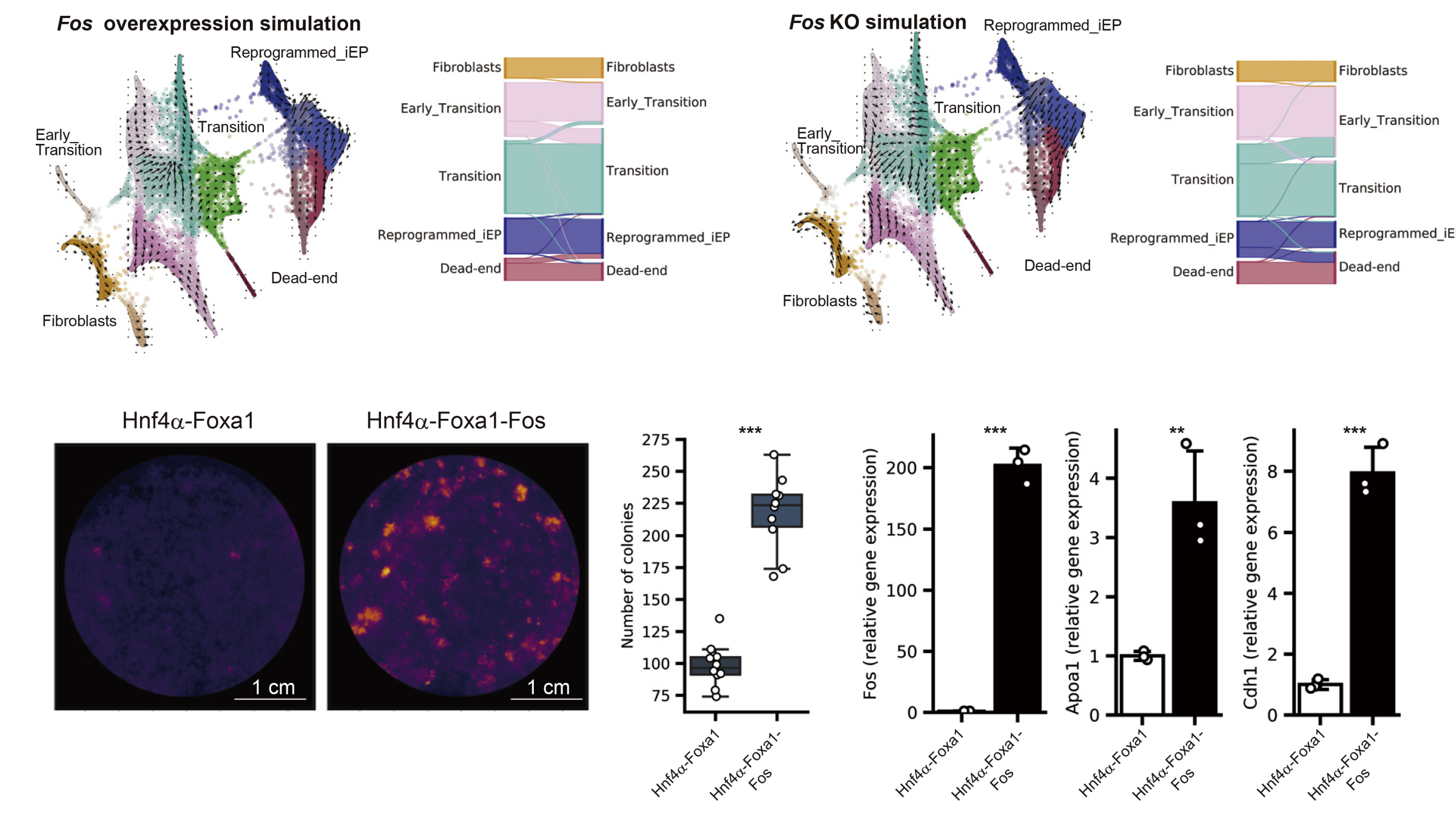
1細胞RNA-seqデータから作成した細胞特異的な遺伝子制御のモデルを構築し、そのモデルを利用したシミュレーションにより細胞のリプログラミングに重要な遺伝子を計算机上で探索する、in silico screening解析を行いました。
本研究は生物データを統合して作成したモデルを利用して細胞設計・生命工学研究を行うという戦略の概念実証の一つです。
Kamimoto, K., et al.,
Gene regulatory network reconfigulation in direct lineage reprogramming.
Stem cell reports 18, 97-112 (2023).
本論文はStem Cell Reportsの2023-2024のTop Cited Paper best 8 にランクインしました。Top-cited stem cell research
Publications
他の論文紹介のページ、論文リストは作成中です。現在全論文リストは下記のGoogle scholarのページより参照願います



